Projekte
Proteomik
Massenspektrometrie
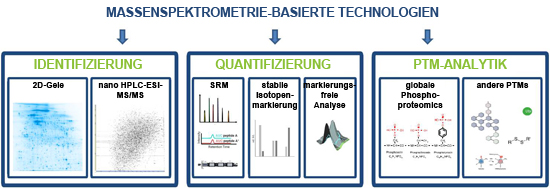
Schlüsseltechnologie für die Identifizierung und Quantifizierung von Proteinen in biologischen Proben ist die biologische Massenspektromtrie. Die Einsatzmöglichkeiten dieser Technik gehen dabei weit über die Identifzierung von Proteinen in 2D-Gel Spots hinaus. Mittels einer sogenannten markierungsfreien Analyse ist es beispielsweise möglich, Proteine aus minimalen Probenmengen ohne den Einsatz von 2D-Gelen auf globaler Ebene zu identifizieren und zu quantifizieren. Das geschieht anhand ihrer Intensitäten im LC-MS Lauf, wobei die verschiedenen Proben direkt miteinander verglichen werden. Eine andere Möglichkeit zur genauen Quantifizierung auf globaler Ebene bietet der Einsatz stabiler schwerer Isotope. So können zwei oder mehr Proben gemischt und im selben Lauf direkt miteinander verglichen werden, da sich die Massen der identischen Proteine in den verschiedenen Proben je nach eingesetzter Isotopenmarkierung unterscheiden. Eine Möglichkeit zur gezielten, sehr genauen und absoluten Quantifizierung einzelner (Kandidaten-) Proteine bietet die SRM-AQUA Methode, bei der ausgewählte synthetische Isotopen-markierte Peptide (Analoga zu nativen Peptiden des Proteins) als interner Standard in einer definierten Menge zur Probe gemischt und dann gezielt im Massenspektrometer detektiert werden.
Zudem können Phosphorylierungen, die in vielen zellulären Prozessen eine essentielle Rolle in der Regulation und Signaltransduktion spielen und krankheitsrelevant sein können, auf globaler Ebene analysiert und quantifiziert werden. Auch andere posttranslationale Modifikationen, wie beispielsweise Acetylierungen oder Glycosylierungen, lassen sich mit massenspektrometrischen Methoden nachweisen.