Projekte
Bioinformatik
Die Bioinformatik stellt das Bindeglied aller anderen Arbeitsgruppen in PURE dar. Die Verbindungen aller im Projekt erstellten Daten aus den anderen Teilbereichen werden hier nicht nur gespeichert, sondern auch im biologischen Kontext verknüpft. Die teilweise komplementären Ansätze eignen sich ideal, um die Informationen, die sich aus den verschiedenen Plattformtechnologien ergänzen, zu einem besseren Allgemeinbild der Erkrankung zusammenzufügen. Die Entwicklung von Algorithmen und Software wird somit zusammen mit der nötigen Hardware übergreifend etabliert und zur Verfügung gestellt. Die hierdurch bessere Klassifizierung von Patienten ergibt unter Verwendung von modernen Methoden der Bioinformatik und Biostatistik die Möglichkeit, die in PURE zu betrachtenden Erkrankungsursachen so individuell zu ermitteln, dass eine personalisierte Medizin durch die projektweite Datenanalyse eine neue Qualität erreicht.
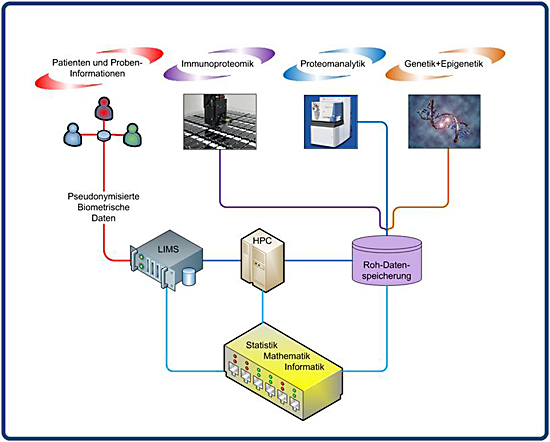
In den Bioinformatik Arbeitsgruppen in PURE werden die Daten der experimentellen und klinischen Arbeitsgruppen analysiert. Diese Analysen beruhen einerseits auf etablierten algorithmischen Ansätzen zur Analyse mikroskopischer, proteomischer, transkriptomischer oder genomischer Daten; andererseits werden neue statistische und mathematische Verfahren zur Plattform-übergreifenden Datenanalyse entwickelt. Neue und verbesserte Verfahren zur quantitativen Analyse mikroskopischer Bilddaten, insbedondere Daten aus multispektralen Bildgebungsverfahren, werden in der Arbeitsgruppe von Prof. Axel Mosig entwickelt und implementiert. Die Gruppe um PD Dr. Martin Eisenacher beschäftigt sich mit der Analyse proteomischer Daten, während Aspekte der epidemiologischen Statistik durch die Gruppe von Prof. Thomas Behrens untersucht werden.
Aus der Konstellation von Experimentalisten und Klinikern innerhalb von PURE entstehen Fragestellungen der Datenanalyse, die nur begrenzt durch existierende Verfahren der Datenanalyse beantwortet werden können. Um die Verfügbarkeit einer breiten Palette von mikroskopischen und proteomischen Daten bei der Analyse voll ausschöpfen zu können, ist es daher unvermeidlich eine Herausforderung an die Bioinformatik, für diese Konstellation maßgeschneiderte Analyseverfahren zu entwickeln. Dies hat zur Entwicklung einer Reihe von Analysemethoden zur Kombination von Daten aus verschiedenen Mikroskopie- oder Proteomik-Plattformen geführt. In zukünftigen Arbeiten sollen diese Arbeiten systematisch weiter geführt werden, so dass für möglichst alle in PURE durchgeführten Studien mit unterschiedlichen Konstellationen parallel verwendeter Mikroskopie- und Proteomik-Plattformen dedizierte Datenanalyseverfahren entwickelt werden.
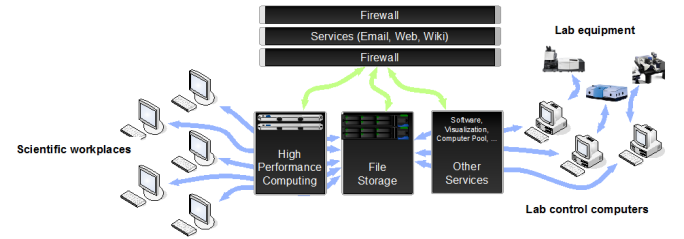
Um gemeinsame Speicherung und Auswertung der Daten aus unterschiedlichen Plattformen zu ermöglichen, ist eine IT Infrastruktur mit entsprechenden Kapazitäten an Datenspeicherung und Rechenleistung erforderlich. Hierzu wurde in den vergangenen Jahren eine IT-Infrastruktur mit mehr als 120 TByte Datenspeicherkapazität sowie Hochleistungsrechner mit mehr als 1500 Prozessor-Kernen aufgebaut.