Projekte
Proteomik
Bioinformatik / Biostatistik
Aufgabe der Bioinformatik ist die Analyse und statistische Auswertung der in den experimentellen Arbeitsgruppen gemessenen Daten. Hierbei werden Ergebnisse ausgewertet, die mit verschiedenen Proteomik–Methoden erzeugt wurden (z. B. mit Methoden der bottom-up Massenspektrometrie, targeted Proteomik, Protein Bio-Chips).
Aufgrund des dominierenden biomedizinischen Kontexts, ist ein häufiges Ziel der bioinformatorischen Auswertung die Identifizierung von Biomarkerkandidaten, die vielversprechend sind für die Früherkennung von Krankheiten. Dies ist auch entscheidend für eine erfolgversprechendere therapeutische Intervention. Die Bioinformatik arbeitet darüber hinaus auch an der Entwicklung neuer Verfahren und Methoden, die an die technische Entwicklung der Messinstrumente (z. B. Verfügbarkeit hochauflösender Massenspektrometer) angepasst sind, um möglichst umfassende und verlässliche Informationen aus den zur Verfügung stehenden Daten zu extrahieren.
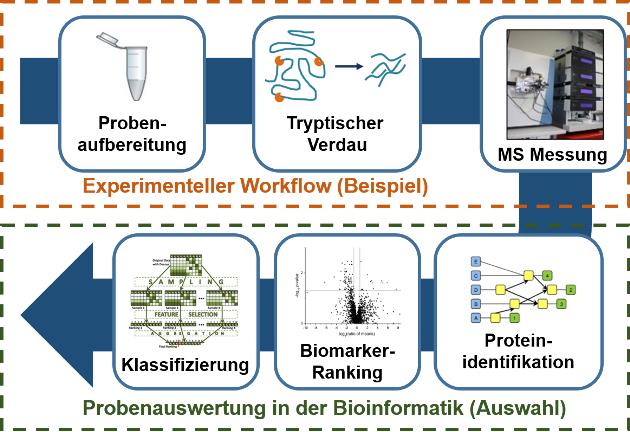
Skizze der Verschränkung des experimentellen Arbeitsablaufs (roter gestrichelter Kasten) mit der nachfolgenden Datenanalyse in der Bioinformatik (grüner gestrichelter Kasten).
Bioinformatische Tools werden von uns in enger Abstimmung mit den experimentell arbeitenden Gruppen ständig weiterentwickelt und neu aufgesetzt, um beispielsweise die Identifizierung von Biomarkerkandidaten bei den verschiedenen Indikationen fortlaufend zu verbessern. Ein wichtiges Element der interdisziplinären Zusammenarbeit ist die gemeinsame, frühzeitige Erarbeitung eines adäquaten Studiendesigns. Wichtige Stichworte in diesem Zusammenhang sind beispielsweise Fallzahlplanung und die Berücksichtigung einer klar formulierten Fragestellung bei der detaillierten Planung der Analyseschritte, die sich an die Messung anschließen.
> Spektrenidentifikation (unter Verwendung mehrerer Suchmaschinen) und Quantifizierung. Die Bioinformatik bietet für diese Aufgabe mehrere, an die verschiedenen Fragestellungen angepasste Auswerte-Workflows an.
> Voll parametrisierbare Proteininferenz aus den ermittelten Peptid–Spektrum–Zuordnungen unter Verwendung des in der Bioinformatik entwickelten Tools PIA (Protein Inference Algorithms).
> Kontrolle der Falsch-positiv–Rate über den sogenannten decoy (Köder)-Ansatz. Die benötigten target/decoy–Datenbanken können mit dem von uns entwickelten Tool DecoyDatabaseBuilder erstellt werden.
> Ranking der Ergebnisse unter gleichzeitiger Berücksichtigung von p-Werten und dem Ausmaß der berechneten Regulation (sog. Fold change). Hierfür stehen in der Bioinformatik entwickelte KNIME Workflows zur Verfügung, die auf R-Skripten basieren.
> Klassifizierungs-Algorithmen (z. B. für die Einordnung neuer Patienten-(Daten) in bestehende Gruppen). Für diese Aufgabe wurde in der Bioinformatik das Tool PAA (Protein Array Analyzer) entwickelt.