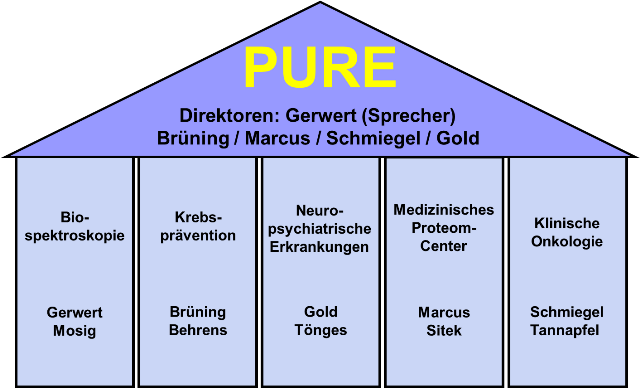
PURE Projects
Biospectroscopy
Stellung in PURE
In der Säule Biospectroscopy werden mit Hilfe modernster vibrationsspektroskopischer Verfahren in Kombination mit moderner bioinformatischer Bildanalyse (Mosig), Gewebe, Zellen, Proteinnetzwerke und Proteine untersucht. Damit können markerfrei und nichtinvasiv krankheitsverursachende Veränderungen der Proteine in Proteinnetzwerken, in der Zelle und im Gewebe mit Hilfe spektraler Biomarker identifiziert werden. Dies ist ein völlig neuer vielversprechender Ansatz über verschiedene Skalen, der jetzt in PURE etabliert wurde.
Laufende Arbeiten
Die spektrale Histopathologie wurde an Darmgewebe in Zusammenarbeit mit Prof. Tannapfel und Prof. Schmiegel etabliert. Damit kann das Gewebe jetzt histopathologisch markerfrei annotiert werden. In derzeitigen Arbeiten wird das „Grading“ des Tumors bestimmt, so dass eine differenzierte Annotation erfolgen kann. Die bisherigen Arbeiten wurden an paraffinierten Schnitten durchgeführt und werden in den derzeitigen Arbeiten auf Frischgewebe erweitert. Neben Darmgewebe wird derzeit Blasengewebe in Zusammenarbeit mit Prof. Tannapfel, Prof. Brüning und Prof. Sitek analysiert. Auch hier kann das Gewebe markerfrei spektral-histopathologisch annotiert werden. Als dritte Entität wird jetzt Lunge in Zusammenarbeit mit Prof. Brüning und Prof. Meyer und der Ruhrlandklinik bearbeitet. Mit Hilfe der Raman Spektroskopie wurden Zellen von Darmkrebszelllinien dreidimensional vermessen und die Verteilung von Kinasehemmern und Antikörpern der EGFR Therapie in der Zelle in Zusammenarbeit mit Prof. Reinacher-Schick und Prof. Schmiegel vermessen. Auch das Therapieansprechen konnte bestimmt werden. Es wurde ebenfalls im Urin nach spektralen Biomarkern gesucht. Vielversprechend sind identifizierte spektrale Biomarker für Blasenkrebs im Harnsediment in Zusammenarbeit mit Brüning. Bei neurodegenerativen Erkrankungen soll mit Hilfe eines hypothesengeleiteten Ansatzes in Zusammenarbeit mit Prof. Wiltfang die Fehlfaltung von Proteinen in Liquor als Früherkennungsmarker entwickelt werden.
Cancer Prevention
Stellung in PURE
Die Säule Krebsprävention untersucht mit einem humanbasierten Ansatz die Signalwege und Mechanismen der Entstehung von Blasen- und Lungenkrebs, um hieraus erfolgreiche Konzepte zur Früherkennung mittels Biomarkern abzuleiten. Dazu legt das Wissenschaftlich-Epidemiologische Studienzentrum unter Berücksichtigung der Empfehlungen der „Guten Epidemiologischen Praxis“ das Fundament für alle PURE-Plattformtechnologien. Hierzu gehören eine gesetzlich vorgeschriebenen ethischen Standards folgende Studiendurchführung, die datenschutzrechtlich gesicherte Erfassung der Studieninformationen, die qualitätsgesicherte Sammlung von Bioproben und die Verwaltung und Bereitstellung der Infrastruktur für eine Bioprobenbank als Basis für die translationale Forschung auf dem Gebiet der Biomarker-gestützten Früherkennung von Blasen- und Lungentumoren. Die Molekulare Tumorbiologie charakterisiert die Proben mittels proteinchemischer, mikroskopischer und genetischer Verfahren und schlägt damit eine Brücke zwischen Geno- und Phänotyp.
Laufende Arbeiten
Im Bereich „Harnblase wurden in Kooperation mit dem Marienhospital Herne sämtliche Voraussetzungen für eine erfolgreiche Gewinnung, Verarbeitung, Transport und Lagerung der Bioproben erfüllt, um qualitativ gesicherte und plattformübergreifende Standards in PURE zu gewährleisten. Das erfolgreich etablierte Datenschutzkonzept wird mit der Ärztekammer Westfalen-Lippe als Treuhänder umgesetzt. Im Bereich „Lunge“ konnte in Kooperation mit der Ruhrlandklinik in Essen erfolgreich ein erstes durch die Deutsche Gesetzliche Unfallversicherung gefördertes Drittmittelprojekt zur Früherkennung von Lungentumoren akquiriert werden. Schließlich konnte im Bereich „Gesunde Kontrollprobanden“ eine Kooperation mit der Heinz-Nixdorf-Recall (HNR)-Studiengruppe in Essen geschlossen werden. Die HNR-Studie ist eine Pilotstudie für die im Aufbau befindliche Nationale Kohorte mit ca. 200.000 Personen.
Neuropsychiatric Diseases
Stellung in PURE
Die Säule Neuropsychiatrische Erkrankungen verfügt über ein klinisches Studienzentrum mit überregionaler Patientenzuweisung mit Schwerpunkt im Bereich Multiple Sklerose und Parkinson-Erkrankungen. Die in diesem Rahmen aufgebaute Biobank mit umfassenden klinischen Datensätzen steht den Mitgliedern der PURE-Initiative zur Verfügung. Proteinbiochemische Forschungsansätze werden mit Daten zu epigenetischen Veränderungen, miRNAs, molekularer und struktureller Bildgebung ergänzt. Die hypothesengesteuerte Suche nach neuen Biomarkern und deren Evaluierung wird komplementiert durch Untersuchungen an Zellkultur-basierten und Tiermodellen der MS und der Parkinson-Erkrankung. Grundlegendes Ziel ist die Entwicklung von Biomarkern zur Verbesserung der Frühdiagnostik, Evaluierung differentieller Erkrankungssubtypen, Graduierung der Erkrankungsprogression sowie des Therapiemonitoring bei MS und Parkinson. Der Bereich Neurodegeneration unter Leitung von Prof. Dr. R. Gold hat in 2015 den Bereich Neuropsychiatrische Erkrankungen (Prof. Dr. J. Wiltfang) ersetzt. Prof. R. Gold wird im Bereich entzündlicher Neurodegeneration von Prof. A. Haghikia und im Bereich primärer Neurodegeneration von Prof. Dr. L. Tönges unterstützt.
Laufende Arbeiten
Die 2015 gegründete Arbeitsgruppe Biobank der medizinischen Fakultät entwickelt derzeit das Konzept einer zentralen Organisation und IT-Struktur einer Biomaterialbank mit dezentraler Lagerung der Biomaterialien. Ethik- und Datenschutzkonzepte werden für die existierenden Biobanken zudem weiter überarbeitet und für zukünftige lokale Biobanken individuell neu entwickelt.
Im longitudinalen Monitoring einer Parkinson-Patientenkohorte wird eine breite klinische Charakterisierung und Bioproben-Phänotypisierung durchgeführt. Dazu wurde die Rekrutierung von Parkinson-Patienten mit vorwiegend idiopathischer Parkinson-Erkrankung im St. Josef-Hospital weiter ausgebaut und Arbeiten des Verbundprojektes „ParkCHIP“ mit dem Medizinischen Proteom Center (MPC) der RUB, der St. Josef- und St. Elisabeth-Hospital gGmbH, Neurologische Universitätsklinik im St. Josef-Hospital Bochum (JOBO), und dem IPA zur Entwicklung eines immunologisch orientierten Biomarker-Chips bei Patienten mit Parkinson-Syndrom werden fortgeführt. Biomarker-Kandidaten zur Differenzierung von Parkinson-Subtypen, Marker zur Krankheitsprogression oder putativ therapeutische Zielstrukturen werden in translationalen Parkinson-Modellen weitergehend untersucht.
Zur Entwicklung von krankheitsdiskriminierenden Biomarkern für Verlaufstypen der MS wurde eine Liquor-Probenbank etabliert, die über 6500 Liquorproben umfasst und weitestgehend von Patienten unserer neuroimmunologischen Ambulanz und auf stationärer Ebene akquiriert werden. In einer longitudinalen Beobachtung werden klinische und paraklinische Daten, wie u. a. MRT und evozierte Potentiale, analysiert, um Surrogatmarker zu entwickeln, die MS-Verlaufstypen und Therapieresponsivität differenzieren können. Darüber hinaus werden Liquor-Proteine als Degenerationsmarker bei MS validiert, die objektivierbar das Ausmaß der Neurodegeneration im Verlauf der MS quantifizieren sollen.
Proteomics
Stellung in PURE
Das Medizinische Proteom-Center ist eines der weltweit führenden Forschungsinstitute im Bereich der klinischen Proteomforschung. Forschungsschwerpunkte sind die Fortentwicklung geeigneter bioanalytischer Methoden für die verbesserte Analyse klinisch relevanter, humaner Proben sowie deren Anwendung bei Krebs-, neurodegenerativen und neuromuskulären Erkrankungen. Durch die innerhalb von PURE finanzierte technologische Plattform können wir bereits mit kleinsten Probemengen, wie sie durch Lasermikrodissektion zu erhalten sind, verwertbare Resultate erzielen. Diese Bioanalytik auf höchstem Niveau wird ergänzt durch eine leistungsfähige Bioinformatikgruppe, die es uns ermöglicht, die anfallenden, außerordentlich umfangreichen Daten der Massenspektrometrie zeitnah auszuwerten.
Laufende Arbeiten
Die stetige Verbesserung der technischen Infrastruktur erlaubt uns, mit unseren massenspektrometrischen und weiteren Analysemethoden an vorderster Front der Bioanalytik mitzuspielen.
Im Bereich der neurodegenerativen Erkrankungen stehen Untersuchungen bei Morbus Parkinson im Fokus, wobei es unser Ziel ist, frühdiagnostische sowie Verlaufsmarkerproteine und dagegen gerichtete Autoimmunantikörper zu identifizieren. Dies bildet die Grundlage für die zukünftige Entwicklung verlaufsmodifizierender oder kurierender Therapieansätze. Proteomstudien bei neuromuskulären Erkrankungen helfen uns, grundlegende pathologische Prozesse besser zu verstehen und Proteinbiomarker für eine verbesserte Diagnostik zu identifizieren.
Im Bereich von Krebserkrankungen stehen zurzeit Untersuchungen bei Blasen-, Leber-, Darm- und Lungenkrebs im Fokus. Mit den Massenspektrometrie-basierten Methoden zu Proteinquantifizierung ist es möglich, Proteomanalysen an Tumorzellen durchzuführen und Krankheits-relevante Proteine zu finden. Diese potentiellen Proteinbiomarker werden anschließend in Körperflüssigkeiten verifiziert, um ihren Einsatz als nicht invasive Biomarker zu testen. Des Weiteren werden Tumor-relevante Signalwege der Zellen untersucht mit dem Ziel, die Tumorgenese auf Proteinebene aufzuklären.
Beide Bereiche werden von einer Bioinformatik-Gruppe unterstützt, die sowohl State-of-the-Art-Auswertungen anwendet, als auch neue Auswertealgorithmen entwickelt, für die es noch keine kommerzielle Lösung gibt.
Der etablierte Workflow kombiniert die Proteingewinnung aus verschiedenen humanen Proben, die Quantifizierung auf Proteinebene sowie eine ausführliche bioinformatische und statistische Auswertung der Daten zur Identifizierung vielversprechender Biomarkerkandidaten. Diese werden an größeren und unabhängigen Kohorten verifiziert.
Clinical Oncology
Stellung in PURE
Eine wesentliche Lösung der Therapieprobleme in der klinischen Onkologie wird angesichts einer zunehmenden Vielfalt von Therapeutika in der Verfügbarkeit von prognostischen und therapieprädiktiven Biomarkern als Voraussetzung für eine personalisierte Medizin gesehen. Für die meisten Bereiche in der klinischen Onkologie ist dieses bislang ein nicht eingelöstes Versprechen, welches eine intensive Forschung zur Identifizierung, Validierung und klinischen Erprobung von Biomarkern erzwingt. In dem von der klinischen Onkologie in PURE gewählten Lösungsansatz konzentrieren wir uns auf Tumoren der Verdauungsorgane (gastroenterologische Onkologie) und Lymphome.
Laufende Arbeiten
Als wichtige Grundlage für unsere Forschungsarbeiten haben wir eine nach aktuellen Standards entwickelte Biomaterialienbank etabliert. Mit Hilfe der Biomaterialien gelang es uns in Kooperation mit der Bioinformatik von PURE, eine Reihe von vielversprechenden Biomarkern für das Kolon- und Pankreaskarzinom bzw. auch für primäre Lymphome des ZNS zu identifizieren, die sowohl für die Prognose, Diagnose aber auch für die Therapie der genannten Karzinomentitäten Anwendung finden können. Das Gesamtkollektiv der zentralen Biomaterialienbank umfasst inzwischen mehr als 2800 Patienten mit liquiden und/oder Gewebeproben und den zugehörigen klinischen (Verlaufs-) Daten. Darüber hinaus wurden Daten von noch etwa 1200 Patienten ohne liquide Proben retrospektiv erfasst. Der Gesamtumfang der liquiden Probensammlung beläuft sich mittlerweile auf mehr als 45.000 Einzelproben (Serum, Plasma, Citratplasma und Blutzellen) sowie auf über 1000 Stuhlproben.
Darüber hinaus haben wir Xenografttumor-Modelle für Pankreas- und Kolonkarzinom etabliert, die für die in vivo Testung aktueller zielgerichteter Therapieschemata eingesetzt werden und bereits zur Identifikation neuer potentiell therapeutisch relevanter Therapietargets geführt haben. Aktuell umfasst die Biobank 200 Patiententumor-abgeleitete Dickdarmkrebs-Xenografttumoren, von denen 90 Tumoren in Kooperation mit der Pathologie mittels NGS auf pathogene Mutationen untersucht sind, sowie 70 Pankreaskarzinom-Xenografttumoren.
Die derzeit laufenden Arbeiten konzentrieren sich auf die Evaluation der klinischen Bedeutung der Liquid Biopsy bei der Therapiesteuerung der metastasierten kolorektalen Karzinome. Aktuell wird das etablierte, patiententumor-abgeleitete Xenograftmodell für den Dickdarmkrebs zum einen für die Optimierung der personalisierten Therapie bei Sekundärresistenz gegenüber einer anti-EGFR Therapie und zum anderen zur Entwicklung neuer zielgerichteter Therapiestrategien für Dickdarmkrebs mit nachgewiesener KRAS-Mutation eingesetzt.
Außerdem arbeiten wir derzeit daran, microRNA-basierte Biomarker im Liquor cerebrospinalis zu identifizieren, um die Differenzierung zwischen primären und sekundären ZNS-Lymphomen zu erlauben. Eine histopathologische oder bildgebende Differenzierung zwischen primären und sekundären ZNS-Lymphomen, die eine klinisch inapparente systemische Manifestation aufweisen, ist derzeit nicht möglich. Da die beiden Erkrankungen unterschiedliche Prognosen aufweisen und unterschiedliche Therapiekonzepte verlangen, ist eine frühe Differenzierung zwischen den beiden Erkrankungen klinisch relevant.
In der Glaskörperflüssigkeit suchen wir nach microRNA-basierten Biomarkern für das primäre vitreoretinale Lymphom, das einen hochmalignen Tumor des Auges darstellt. Da der bisherige Goldstandard eine Fehlerquote von bis zu 30% bei der Abgrenzung zur Entzündung aufweist, besteht derzeit ein dringender Bedarf nach verbesserter Diagnostik.
Bioinformatics
Die in PURE gewonnen Daten werden plattformübergreifend statistisch, mathematisch und bioinformatisch analysiert. Ausgehend davon werden neue und verbesserte Methoden und Algorithmen, insbesondere zur Analyse multispektraler Bilddaten, entwickelt und implementiert. Diese Kombination von Anwendung und Entwicklung auch grundlegend neuer Algorithmen ist nur durch den plattformübergreifenden Charakter der Bioinformatik in PURE möglich und vereint alle Komponenten als Querschnittsaufgabe. Dazu gehört auch die epidemiologische Interpretation. Die zu erstellenden epidemiologischen Bibliotheken und Bioinformatik-Softwarepakete werden ein Alleinstellungsmerkmal sein.
Additional Projects
-
BIO.NRW PROFILE
Prädikative Biomarker und Drug targets für das individualisierte Management von Lebererkrankungen. -
Cancer-Sys / HNPCC-Sys
Eine Initiative des Bundesministeriums für Bildung und Forschung (BMBF)zur Systembiologie des erblichen Darmkrebses. -
Charakterisierung des Interaktoms verschiedener Spleißvarianten der ß2-Untereinheit von spannungsabhängigen L-Typ Kalziumkanälen in Kardiomyozyten.
-
http://www.krebsimleben.de/tl_files/pdf/Colon-Resist-Net.pdf
Projekt zur Optimierung der individualisierten Dickdarmkrebstherapie beim Vorliegen einer anti-EGFR-Therapie Sekundärresistenz mit Hilfe eines Avatar-Modells. -
DPG - Patientenregister
In einer Kooperationsstudie mit der Arbeitsgruppe atypische Parkinson-Syndrome der Deutschen Parkinson Gesellschaft (DPG) wird derzeit ein spezialisiertes nationales Patientenregister aufgebaut. Ziel des Vorhabens ist die Beschreibung des natürlichen Krankheitsverlaufs im möglichst frühen Erkrankungsstadium der u. a. der Progressiven Supranukleären Parese (PSP), insbesondere von deren verschiedenen klinischen Prädominanztypen, um die frühe klinische Diagnose zu optimieren, diagnostisch und prognostisch relevante Krankheitsparameter zu identifizieren, neuartige diagnostischen Modalitäten und Progressions-Messparameter (z. B. MRT Morphometrie) zu evaluieren und eine solide prospektive longitudinale Datenbasis für die Planung zukünftiger klinischer Therapiestudien zu generieren. -
Einsatz proteinanalytischer Verfahren zur (Früh-)Diagnose asbestassoziierter Lungen- und Pleuratumoren
Lungentumoren früher erkennen und so die Heilungschancen verbessern – das ist das Ziel des von der Deutschen Gesetzlichen Unfallversicherung (DGUV) geförderten Forschungsprojekts. Zu diesem Zweck kooperieren hier Forscher des Europäischen Proteinforschungsinstituts PURE, des Universitätsklinikums Köln/Bonn und des Instituts für Prävention und Arbeitsmedizin der DGUV. -
Einsatz proteinanalytischer Verfahren zur (Früh-)Diagnose von Blasenkrebs (UroFollow)
Zu diesem Zweck kooperieren hier Forscher des Europäischen Proteinforschungsinstituts PURE, der Schön Klinik Nürnberg Fürth und der Universitätsklinika Bochum, Erlangen Rostock und Tübingen sowie des Instituts für Prävention und Arbeitsmedizin der DGUV. -
Familiärer Darmkrebs
Ein Verbundprojekt der Deutschen Krebshilfe -
FOR 1228: Molecular Pathogenesis of Myofibrillar Myopathies
-
i:DSem – Integrative Datensemantik in der Systemmedizin
Eine Initiative des Bundesministeriums für Bildung und Forschung (BMBF) zur integrativen Datensemantik in der Systemmedizin. -
Kinetics for Drug Discovery (K4DD)
Ein im Rahmen der Europäischen Innovative Medicines Initiative (IMI)gefördertes Projekt zur Untersuchung der kinetischen Aspekte der Interaktion zwischen Wirkstoff und Drug-Target für die in vivo Effizienz von Arzneimitteln. -
Mechanismen der neuroprotektiven Aktivität der E3-Ubiquitin-Ligase Parkin
- NaDiNe Nanosystems for Early Diagnosis of Neurodegenerative Diseases ist ein Projekt zur Entwicklung neuer Verfahren zur frühen Diagnose neurodegenerativer Erkrankungen
- Neuroallianz Eine Forschungskooperation im Rahmen der Pharma-Initiative Deutschland - Innovative Therapeutika und Diagnostika für neurodegenerative Erkrankungen.
-
NISCI – Antibodies against Nogo
A to enhance regeneration and functional recovery after acute spinal cord injury a multicenter European clinical proof of concept Trial. -
OsteoSys
Osteoporose – eine Volkserkrankung im Kontext von kardiovaskulärer Komplikationen und chronischer Inflammation: systemmedizinischer Ansatz zur personalisierten Therapie. - PharmaCog "Prediction of cognitive properties of new drug candidates for neurodegenerative diseases in early clinical development" (PharmaCog) ist ein im Rahmen der Europäischen Innovative Medicines Initiative (IMI)gefördertes Projekt zur Untersuchung potentieller Drug-Targets und zur schnelleren Entwicklung und damit der schnelleren Zulassung neuer Medikamente für neurodegenerative Erkrankungen.
-
Projekt zur Isolierung von spezifischen Neuronenpopulationen und Gliazellen aus humaner Substantia nigra.
- RESIST-Net Im Mai 2016 hat das unter Bochumer Federführung gegründete, nationale Netzwerk RESIST-Net (mit DKFZ & NCT-Heidelberg sowie der LMU München) seine Arbeit aufgenommen. Ziel des Projekts ist es, aufbauend auf unseren Vorarbeiten zu klären, wie zuverlässig patiententumor-abgeleitete Xenograftmodelle in der Lage sind, die anti-EGFR Sekundärresistenz, wie sie sich im Patienten entwickelt, abzubilden, und ob patiententumor-abgeleitete Xenograftmodelle einen effektiven Ansatz darstellen, die individualisierte Therapie für Patienten zu optimieren. Schließlich soll geklärt werden, ob der molekulare Datensatz, der in diesen Versuchen erhoben wird, dafür eingesetzt werden kann, ein bioinformatisches Modell zur Vorhersage der Sekundärresistenzursachen und der daraus resultierenden Therapiemöglichkeiten zu etablieren.
-
Service Center ‘Bioinformatics for Proteomics – BioInfra.Prot
Das Service Center stellt eigens entwickelte Software für Proteomanalysen zur Verfügung und bemüht sich um Datenstandardisierung und Datenkonvertierung. - SFB Transregio 60 Interaktion von Viren mit Zellen des Immunsystems bei persistierenden Virusinfektionen: Grundlagen für Immuntherapie und Impfungen.